 Felles avstamning? av Casey Luskin -Bokanbefaling av 'Theistic Evolution'
Felles avstamning? av Casey Luskin -Bokanbefaling av 'Theistic Evolution'
1. Teistisk evolusjon omfavner felles avstamning
Fr. Collins argumenterer i 'The Language of God' at "konklusjonen om en felles stamfar for mennesker og mus er i realiteten uunngåelig." Noe lignende sier andre teistiske evolusjonister som G. Gilberson, Dennis Venema og Robert Asher. De to førstnevnte sammenligner de som betviler dette med geosentrikere. (7)
Før vi undersøker bevis om dette, er det viktig å legge merke til at felles avstamning kan være feilaktig eller holde stikk på ulike taksonomiske nivåer. Således kan universell felles avstamning være feilaktig, mens felles avstamning kan holde stikk på lavere taksonomiske nivåer som f.eks. familie eller orden. Felles avstamning må til slutt vurderes ut fra en sak til sak vurdering, avhengig av bevis innenfor den bestemte gruppa som studeres. Her skal vi evaluere saken for universell felles avstamning, slik den vanligvis fremstilles i lærebøker og populærbøker av teistiske evolusjonister og evolusjons-talsmenn.
Av plasshensyn må vi her begrense oss til å betrakte DNA og anatomiske likheter som grunnlag for problemstillingen. La oss se hva som kan være alternativ til felles avstamning. Det kan være i forhold til blinde og tilfeldige naturprosesser, rene tilfeldigheter eller så kan det være at genetiske likheter kan skyldes 'gjenbruk av funksjonelle komponenter' i ulike taksonomiske klasser. Felles design er dermed en levedyktig forklaring på funksjonelle likheter blant bio-molekyler som er funnet i ulike typer av organismer. Som forfatterne av en artikkel fant: "atskillige kjerne-komponenter til det bakterielle replikasjons-maskineriet er enten ikke-relatert eller bare fjernt relaterte til funksjonelt ekvivalente komponenter i arkea/eukaryote replikasjons-apparatet. Noe som ledet dem til å foreslå: "DNA replikasjon skjedde sannsynlig uavhengig av hverandre i bakterielle og arkeisk/eukaryote avstamnings-linjene. (55) En annen artikkel sammenlignet genomet med 1000 ulike prokaryote organismer, og fant at "av de 1000 genom tilgjengelig, ble ikke et eneste protein bevart over alle genom." (56)
Men det er ikke bare likheter mellom bio-molekyler, som gjør at biologer hevder at demonstrerer universell felles avstamning. De hevder ofte at mønstre av lignende nukleotider og aminosyre-sekvenser for gener og proteiner, tillater at organismer kan organiseres i et fylogenetisk 'livets tre' (Bilde 2), som viser evolusjonære relasjoner mellom alle levende organismer. (57) Det var eneste illustrasjon, og har blitt det mest berømte ikon som representerer teorien. Men eksisterer 'livets tre'?
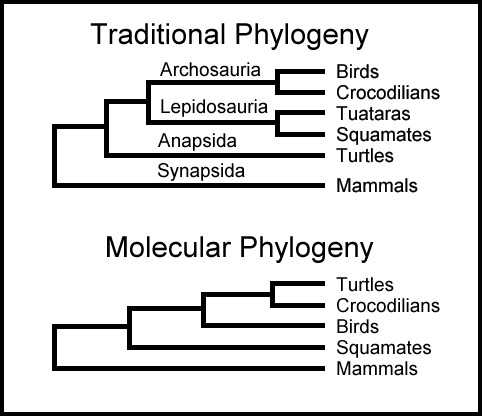 På 1960-tallet, snart etter avdekkingen av den genetiske kode, var det forventet at fylogenetiske trær ville bekrefte formodningen om felles avstamning, som evolusjons-biologer opptatt av morfologi (fysiske trekk ved organismer) holdt seg med. De erklærte at "Om de to fylogenetiske trærne -ut fra morfologi og DNA-sekvenser, i hovedsak er i harmoni i forhold til forgrening, vil det skaffe det beste bevis på realiteten ved makro-evolusjon." (58) Formodentlig hvis denne forutsigelsen feilet, og det var skarpe konflikter mellom trærne som ble dannet ut fra ulike datakilder, så ville det forsyne et tvingende motbevis til makroevolusjon. I håp om å få bekreftet denne forutsigelsen startet biologer å sekvensiere gener fra alle slags levende organismer. I 1990-årene førte det til en oppdagelse som forvirret evolusjonære biologer: Livet delte seg i tre domener, som ikke kunne forenes i et pent tre-liknende mønster. Således klagde den prominente biokjemiker W. Ford Doolittle: "Molekylære fylogenier vil ha feilet i å finne det 'sanne treet', ikke fordi deres metoder er utilstrekkelige eller fordi de har valgt feil gener, men fordi livets historie ikke skikkelig kan bli representert ved et tre." (59) Han forklarte at for mange biologer, "er det det samme som å ha feilet i oppgaven Darwin satte for oss: å skissere denunike strukturen til livets tre." (59)
På 1960-tallet, snart etter avdekkingen av den genetiske kode, var det forventet at fylogenetiske trær ville bekrefte formodningen om felles avstamning, som evolusjons-biologer opptatt av morfologi (fysiske trekk ved organismer) holdt seg med. De erklærte at "Om de to fylogenetiske trærne -ut fra morfologi og DNA-sekvenser, i hovedsak er i harmoni i forhold til forgrening, vil det skaffe det beste bevis på realiteten ved makro-evolusjon." (58) Formodentlig hvis denne forutsigelsen feilet, og det var skarpe konflikter mellom trærne som ble dannet ut fra ulike datakilder, så ville det forsyne et tvingende motbevis til makroevolusjon. I håp om å få bekreftet denne forutsigelsen startet biologer å sekvensiere gener fra alle slags levende organismer. I 1990-årene førte det til en oppdagelse som forvirret evolusjonære biologer: Livet delte seg i tre domener, som ikke kunne forenes i et pent tre-liknende mønster. Således klagde den prominente biokjemiker W. Ford Doolittle: "Molekylære fylogenier vil ha feilet i å finne det 'sanne treet', ikke fordi deres metoder er utilstrekkelige eller fordi de har valgt feil gener, men fordi livets historie ikke skikkelig kan bli representert ved et tre." (59) Han forklarte at for mange biologer, "er det det samme som å ha feilet i oppgaven Darwin satte for oss: å skissere denunike strukturen til livets tre." (59)
Hovedproblemet er at ett gen fører til en versjon av 'livets tre,' men ett annet gen fører til et helt annet livets tre. Det som ser ut til å bekrefte et nært evolusjonært slektskap i ett tilfelle, gjør det ikke i et annet". For å si det på annen måte: "biologisk likhet dukker vedvarende opp på steder der det ikke var forutsatt ved felles avstamning, og fører til konflikt mellom fylogenetiske trær. Når det er konflikt mellom to fylogenetiske trær, så må minst ett være feil. Og om ett tre er feil, hvordan vet vi at ikke begge er feilaktige?
Bilde X. Eks. på molekylær vs. tradisjonell fylogeni
Tallrike tekniske artikler har bekreftet hyppigheten av fylogenetiske trær mellom ulike taksonomiske grupper. I 1998, observerte en studie i 'Genome Research' at "ulike proteiner, gir ulike fylogenetiske trær." (61) En artikkel fra 2009 i 'Trends in Ecology and Evolution' erkjente: "evolusjonære trær fra ulike gener har ofte konfliktfylte forgreningsmønstre." (62) En artikkel fra 2013 i 'Trends in Genetics' rapporterte at "dess mer vi lærer om genomer dess mindre tre-like finner vi at deres evolusjonære historie er." (63) Kanskje den mest ærlige innrømmelsen kom i en artikkel fra 2009 i New Scientist, med tittelen "Why Darwin was wrong about the Tree of Life." (64) Det siterte forsker Eric Bapteste som innrømte at " den hellige gral var å bygge livets tre, men i dag ligger det prosjektet sønder knust i biter, slaktet av negative bevis. " I følge artikkelen, "argumenterer mange biologer nå at tre-konseptet er foreldet og trenger å bli forkastet." Artikkelen gjenga resultatene av en studie av M. Syvanen, som sammenliknet to tusen gener over seks dyre-rekker: "I teorien skulle han være i stand til å benytte gensekvenser til å konstruere evolusjonære trær, som viste relasjonene mellom de 6 dyrerekkene. Han feilet. Problemet var at ulike gener fortalte motsetningsfylte evolusjonære historier".
Syvannen forklarte fyndig problemet: "Vi har nettopp utslettet livets tre. Det er ikke et tre lenger, det er en helt ulik topologi. Hva ville Darwin ha gjort ut av det?" Tydeligvis kolliderte ofte molekylbaserte trær med hverandre. Men hva med forutsigelsen (Pauling og Zuckerkandl) at molekylbaserte trær ville matche de som ble dannet ut fra morfologi? En oversikts-artikkel i Nature, med tittelen: "Bones, Molecules or Both?" forklarte at: "Evolusjonære trær som ble konstruert ved å studere biologiske molekyler, ligner ofte ikke på dem som ble konstruert ut fra morfologi, " og innrømte at: " slag mellom molekyler og morfologi blir utkjempet over hele livets tre." (65)
Et klassisk eksempel involverer forsøk på å konstruere et fylogenetisk tre i dyrerekkene. Tradisjonelt er mange dyrerekker blitt gruppert i henhold til hvorvidt de har et sentralt kropps-hulrom (coelom). Men gruppering ut fra molekylære data, motsa den grupperingen, og plasserte i stedet organismer som var meget ulike morfologisk (rundormer og leddyr) meget nært hverandre. En artikkel i Nature rapporterte hvor uventet denne grupperingen var "Tatt i betraktning de store ulikhetene i morfologi, embryo-ulikheter og livshistorier hos de aktuelle dyrene, var det initialt overraskende at ribosomale RNA-treet skulle gruppere dem sammen. " (66) Andre fundamentale dyrekarakteristika, slik som symmetri og tidlige utviklingsprosesser, gir også mønstre av konfliktfylte trær i utbytte.
 Høyere oppe i livets tre, vedvarer konflikter. I 2014 førte sekvensering av ulike genom fra fugl til et uventet resultat at mange typer av fugl, som en tidligere trodde var nært beslektet, utviklet sin gruppes bestemmende trekk konvergent. (68) Som Nature uttrykte det: "livets tre for fugler er blitt omtegnet." (69) Problemet var igjen, at etter genom-data var sekvensiert og forstått, at mange grunnleggende trekk ved og livsstil hos fugl ikke lenger passet inn i et nøstet hierarki. Som et siste eks: En artikkel fra 2013 erkjente problemene en møtte når en skulle forsøke å forsone konfliktfylte versjoner av pattedyr-livstreet: -Å løsrive roten til evolusjonstreet for placenta-pattedyr, har nesten vært en umulig oppgave. Den gode nyheten er at bare tre muligheter er seriøst vurdert. Den dårlige nyheten er at alle tre muligheter er seriøst vurdert. Paleontologer favoriserer en rot med dovendyr og maurslukere (Xenarthra), mens molekylære evolusjonister har favorisert de to andre mulige røttene i form av klippegrevlinger, børstesvin (Afrotheria) og Atlantogenata (Afrotheria + Xenarthra). Nå har to grupper av forskere nøye gått gjennom det størst tilgjengelige genom-datasett om temaet, og har kommet til divergerende konklusjoner... Unødvendig å si, så trengs mer forskning. (70)
Høyere oppe i livets tre, vedvarer konflikter. I 2014 førte sekvensering av ulike genom fra fugl til et uventet resultat at mange typer av fugl, som en tidligere trodde var nært beslektet, utviklet sin gruppes bestemmende trekk konvergent. (68) Som Nature uttrykte det: "livets tre for fugler er blitt omtegnet." (69) Problemet var igjen, at etter genom-data var sekvensiert og forstått, at mange grunnleggende trekk ved og livsstil hos fugl ikke lenger passet inn i et nøstet hierarki. Som et siste eks: En artikkel fra 2013 erkjente problemene en møtte når en skulle forsøke å forsone konfliktfylte versjoner av pattedyr-livstreet: -Å løsrive roten til evolusjonstreet for placenta-pattedyr, har nesten vært en umulig oppgave. Den gode nyheten er at bare tre muligheter er seriøst vurdert. Den dårlige nyheten er at alle tre muligheter er seriøst vurdert. Paleontologer favoriserer en rot med dovendyr og maurslukere (Xenarthra), mens molekylære evolusjonister har favorisert de to andre mulige røttene i form av klippegrevlinger, børstesvin (Afrotheria) og Atlantogenata (Afrotheria + Xenarthra). Nå har to grupper av forskere nøye gått gjennom det størst tilgjengelige genom-datasett om temaet, og har kommet til divergerende konklusjoner... Unødvendig å si, så trengs mer forskning. (70)
Men løser alltid 'mer forskning' problemet? (71) En artikkel fra 2012 bemerket at "fylogenetiske konflikter er vanlige, og oftere regelen enn unntaket," fordi "inkongruens mellom fylogenier oppnådd ved morfologiske vs. molekylære analyser, og mellom trær basert på ulike sub-sett av molekylære analyser, og mellom trær basert på ulike sub-sett av molekylære sekvenser, har blitt vedvarende i det data sett har økt raskt om både egenskaper og arter." (72)
Bilde 3. Cladogram med fuglers plassering
I alle fall har disse hyppige ulikhetene mellom molekylære og morfologisk-baserte trær, og mellom ulike molekylære baserte trær, har fått noen forskere til å konkludere at forutsigelsen til Zuckerkandl og Pauling var fundamentalt feilaktig. En artikkel i journalen 'Biological Theory' forklarte: "Molekylær systematikk basert på formodningen, først uttalt av Zuckerkandl og Pauling, at graden av likhet reflekterer graden av slektskap. Denne formodningen stammer fra å fortolke molekylær likhet (eller ulikhet) mellom dyrerekker i kontekst av en darwinistisk modell med kontinuerlig og gradvis endring. Gjennomsyn av historien med molekylær systematikk og dens krav i kontekst av molekylær biologi åpenbarer at det ikke er noen basis for den 'molekylære formodningen' (73).
For å si det på en annen måte, konflikter mellom molekylære og morfologiske trær, utfordrer seriøst felles avstamning, og .. underminerer metodene som benyttes for å slutte til det. Men hva med de 'lykkelige tilfellene' der molekylbaserte trær viser noe kongruens med mer fylologi-baserte trær. Gir de noen form for spesielle bevis for felles avstamning? Nei ikke i det hele. Husk at morfologiske trær er basert på å sammenlikne likheter mellom anatomiske trekk mellom ulike organismer. Men om anatomiske trekk dannes av gensekvenser i DNA, så følger at organismer med lik anatomi typisk vil ha mer likt DNA. Ut fra denne gen-morfologiske linken, trenger en ikke ta i betraktning felles avstamning for å forutsi at de kan vise noe samsvar. Og begge typer av samsvar kan bli forklart ved felles design like så bra som felles avstamning.
Referanser:
55. D.D. Leipe et al. "Did DNA Replication Evolve Twice Independently?" Nucleid Acids Research 27 (1999): 3389-3401.
56. K. Lagesen et al, "Genome Update the 1000th Genome -A cautionary Tale," Microbiology 156 (Mars, 2010) 603-608
58. E. Zuckerkandl og L. Pauling, "Evolutionary Divergences and Convergence in Proteins," in Evolving Genes and Proteins: A Symposium Held at the Institute of Microbiology of Rutgers, The State University, red. V. Bryson og H. J. Vogel (New York: Academic Press, 19065), 101.
59. W.Ford Doolittle, "Phylogenetic Classification and the Universal Tree," Science 284 (Juni 25, 1999), 2124-2128.
61. A. R. Mushegian et al, "Large-Scale Taxonomic Profiling of Eucaryotic Model Organisms: A Comparison of Orthologous Proteins Encoded by the Human, Fly, Nematode, and Yeast Genomes,"Genome Research (1998): 590-598.
62. J.H. Degnan og N.A. Rosenberg, "Gene Tree Discordance, Pylogenetic Inference and the Multispecies Coalescent," Trends in Ecology and Evolution 24 (2009); 332-340.
63. E. Bapteste et al. "Networks Expanding Evolutionary Thinking," Trends in Genetics 29 (Aug. 2013); 439-441
64. Gr. Lawton, "Why Darwin was Wrong about the Tree of Life," New Scientist (Jan. 21, 2009): 34-39.
65. Tr. Gura, "Bones, Molecules or Both?" Nature 406 (Juli, 2000), s.230-233.
66. A. M. A. Aguinaldo et al, "Evidence for a Clade of Nematodes, Arthropods, and other Moulting Animals," Nature 387, (May 29, 1997): 489-493
68. E. D. Jarvis et al, "Whole Genome Analyses Resolve Early Branches in the Tree of Life of Modern Birds," Science 346, (Dec. 12, 2014): 1320-1331
69. E. Callaway, "Flock of Geneticists Redraw Bird Family Tree," Nature 516 (Des. 11, 2014): 297.
70. E. C. Teeling et al, "Making the Impossible Possible: Rooting the Tree of Placental Mammals," Molecular Biology and Evolution (2013)
71. J.E. Tarver et al, "The Interrelationalships of Placental Mammals and the Limits of Phylogenetic Inference," Genetic Biology and Evolution 8 (2006): 330-344.
72. L. M. Davalos et al, "Understanding Phylogenetic Incongruence: Lessons from Phyllostomid Bats," Biological Reviews of the Cambridge Philosophical Society 87 (2012): 991-1024.
73. J. H. Schwarz et al, "Do Molecular Clocks run at All? A Critique of Molecular Systematics," Biological Theory 1 (Des. 2006): 357-371
Oversettelse og bilder ved Asbjørn E. Lund
 Felles avstamning? av Casey Luskin -Bokanbefaling av 'Theistic Evolution'
Felles avstamning? av Casey Luskin -Bokanbefaling av 'Theistic Evolution' Felles avstamning? av Casey Luskin -Bokanbefaling av 'Theistic Evolution'
Felles avstamning? av Casey Luskin -Bokanbefaling av 'Theistic Evolution' På 1960-tallet, snart etter avdekkingen av den genetiske kode, var det forventet at fylogenetiske trær ville bekrefte formodningen om felles avstamning, som evolusjons-biologer opptatt av morfologi (fysiske trekk ved organismer) holdt seg med. De erklærte at "Om de to fylogenetiske trærne -ut fra morfologi og DNA-sekvenser, i hovedsak er i harmoni i forhold til forgrening, vil det skaffe det beste bevis på realiteten ved makro-evolusjon." (58) Formodentlig hvis denne forutsigelsen feilet, og det var skarpe konflikter mellom trærne som ble dannet ut fra ulike datakilder, så ville det forsyne et tvingende motbevis til makroevolusjon. I håp om å få bekreftet denne forutsigelsen startet biologer å sekvensiere gener fra alle slags levende organismer. I 1990-årene førte det til en oppdagelse som forvirret evolusjonære biologer: Livet delte seg i tre domener, som ikke kunne forenes i et pent tre-liknende mønster. Således klagde den prominente biokjemiker W. Ford Doolittle: "Molekylære fylogenier vil ha feilet i å finne det 'sanne treet', ikke fordi deres metoder er utilstrekkelige eller fordi de har valgt feil gener, men fordi livets historie ikke skikkelig kan bli representert ved et tre." (59) Han forklarte at for mange biologer, "er det det samme som å ha feilet i oppgaven Darwin satte for oss: å skissere denunike strukturen til livets tre." (59)
På 1960-tallet, snart etter avdekkingen av den genetiske kode, var det forventet at fylogenetiske trær ville bekrefte formodningen om felles avstamning, som evolusjons-biologer opptatt av morfologi (fysiske trekk ved organismer) holdt seg med. De erklærte at "Om de to fylogenetiske trærne -ut fra morfologi og DNA-sekvenser, i hovedsak er i harmoni i forhold til forgrening, vil det skaffe det beste bevis på realiteten ved makro-evolusjon." (58) Formodentlig hvis denne forutsigelsen feilet, og det var skarpe konflikter mellom trærne som ble dannet ut fra ulike datakilder, så ville det forsyne et tvingende motbevis til makroevolusjon. I håp om å få bekreftet denne forutsigelsen startet biologer å sekvensiere gener fra alle slags levende organismer. I 1990-årene førte det til en oppdagelse som forvirret evolusjonære biologer: Livet delte seg i tre domener, som ikke kunne forenes i et pent tre-liknende mønster. Således klagde den prominente biokjemiker W. Ford Doolittle: "Molekylære fylogenier vil ha feilet i å finne det 'sanne treet', ikke fordi deres metoder er utilstrekkelige eller fordi de har valgt feil gener, men fordi livets historie ikke skikkelig kan bli representert ved et tre." (59) Han forklarte at for mange biologer, "er det det samme som å ha feilet i oppgaven Darwin satte for oss: å skissere denunike strukturen til livets tre." (59) Høyere oppe i livets tre, vedvarer konflikter. I 2014 førte sekvensering av ulike genom fra fugl til et uventet resultat at mange typer av fugl, som en tidligere trodde var nært beslektet, utviklet sin gruppes bestemmende trekk konvergent. (68) Som Nature uttrykte det: "livets tre for fugler er blitt omtegnet." (69) Problemet var igjen, at etter genom-data var sekvensiert og forstått, at mange grunnleggende trekk ved og livsstil hos fugl ikke lenger passet inn i et nøstet hierarki. Som et siste eks: En artikkel fra 2013 erkjente problemene en møtte når en skulle forsøke å forsone konfliktfylte versjoner av pattedyr-livstreet: -Å løsrive roten til evolusjonstreet for placenta-pattedyr, har nesten vært en umulig oppgave. Den gode nyheten er at bare tre muligheter er seriøst vurdert. Den dårlige nyheten er at alle tre muligheter er seriøst vurdert. Paleontologer favoriserer en rot med dovendyr og maurslukere (Xenarthra), mens molekylære evolusjonister har favorisert de to andre mulige røttene i form av klippegrevlinger, børstesvin (Afrotheria) og Atlantogenata (Afrotheria + Xenarthra). Nå har to grupper av forskere nøye gått gjennom det størst tilgjengelige genom-datasett om temaet, og har kommet til divergerende konklusjoner... Unødvendig å si, så trengs mer forskning. (70)
Høyere oppe i livets tre, vedvarer konflikter. I 2014 førte sekvensering av ulike genom fra fugl til et uventet resultat at mange typer av fugl, som en tidligere trodde var nært beslektet, utviklet sin gruppes bestemmende trekk konvergent. (68) Som Nature uttrykte det: "livets tre for fugler er blitt omtegnet." (69) Problemet var igjen, at etter genom-data var sekvensiert og forstått, at mange grunnleggende trekk ved og livsstil hos fugl ikke lenger passet inn i et nøstet hierarki. Som et siste eks: En artikkel fra 2013 erkjente problemene en møtte når en skulle forsøke å forsone konfliktfylte versjoner av pattedyr-livstreet: -Å løsrive roten til evolusjonstreet for placenta-pattedyr, har nesten vært en umulig oppgave. Den gode nyheten er at bare tre muligheter er seriøst vurdert. Den dårlige nyheten er at alle tre muligheter er seriøst vurdert. Paleontologer favoriserer en rot med dovendyr og maurslukere (Xenarthra), mens molekylære evolusjonister har favorisert de to andre mulige røttene i form av klippegrevlinger, børstesvin (Afrotheria) og Atlantogenata (Afrotheria + Xenarthra). Nå har to grupper av forskere nøye gått gjennom det størst tilgjengelige genom-datasett om temaet, og har kommet til divergerende konklusjoner... Unødvendig å si, så trengs mer forskning. (70)